医学图像分割框架DeepMedSeg
一个用于的开源框架,支持处理和医学数据,并集成了多种流行的分割网络架构。本项目旨在简化研究人员在医学图像分割领域的开发和实验过程,具有模块化、可扩展性强的特点,能够快速定制和测试各种分割模型。集成模型 Swin UNet、MissFormer、TransUNet、Swin UNetr、UNETR、nnUNet、nnFormer。
分享一个用于医学图像分割 的开源框架,支持处理 2D 和 3D 医学数据,并集成了多种流行的分割网络架构。本项目旨在简化研究人员在医学图像分割领域的开发和实验过程,具有模块化、可扩展性强的特点,能够快速定制和测试各种分割模型。
集成模型 Swin UNet、MissFormer、TransUNet、Swin UNetr、UNETR、nnUNet、nnFormer
该框架支持的主要功能包括:
- 2D 与 3D 医学数据的处理,如CT、MRI、自然图片数据等。
- 流行的分割网络集成:包括 U-Net、3D U-Net、Attention U-Net、ResUNet、Swin UNet、MissFormer、TransUNet、Swin UNetr、UNETR、nnUNet、nnFormer 等(目前正在拓展中)。
- 高扩展性:可以轻松集成新模型和数据处理方法。
- 完整的训练与测试流程,包括数据预处理、训练、验证、评估等模块。
目录结构
DeepMedSeg/
├── 2d/ # 2D医学图像分割部分
│ ├── data/ # 存放2D切片数据
│ ├── networks/ # 2D分割模型
│ ├── other_networks/ # 其他扩展的网络
│ ├── utils/ # 2D工具函数
│ ├── test.py # 2D分割的测试代码
│ ├── tester.py
│ ├── trainer.py
│ └── train.py # 2D分割的训练代码
├── 3d/ # 3D医学图像分割部分
│ ├── lib/ # 存放3D分割相关的库
│ ├── networks/ # 3D分割模型
│ ├── dataset_AMOS.json # AMOS数据集配置文件
│ ├── dataset_FeTA.json # FeTA数据集配置文件
│ ├── dataset_FLARE.json # FLARE数据集配置文件
│ ├── load_datasets_transforms.py # 数据加载及增强
│ ├── main_finetune.py # 3D模型微调代码
│ ├── main_train.py # 3D模型训练代码
│ ├── test_seg.py # 3D分割测试代码
├── README.md # 项目说明文件
├── requirements.txt # Python依赖包文件
└── data # 训练数据
安装指南
环境依赖
请确保您的系统安装了以下依赖:
- Python 3.7+
- PyTorch 1.8+
- torchvision
- numpy
- matplotlib
- SimpleITK
- scikit-learn
- albumentations
安装步骤
-
克隆项目到本地:
git clone https://github.com/your-username/DeepMedSeg.git cd DeepMedSeg
-
安装依赖:
pip install -r requirements.txt
数据集准备
项目支持多个公开医学数据集,支持对3D数据的切片
Synapse、AMOS、FLARE
此项目支持以上数据集的二维分割与三维分割,二维分割3D数据的深度切片
将以上数据集按照如下方式组织以支持三维分割
root_dir/
├── imagesTr
├── labelsTr
├── imagesVal
├── labelsVal
├── imagesTs
对于二维分割 ,运行以下命令进行预处理3D数据(以Synapse数据集为例):
cd data/Synapse python Slice_Synapse.py
此后会得到切片后的2D数据,请按照以下结构组织切片数据:
data/synapse
├── Synapse/synapse_2d/
│ ├── train_npz_new/
│ ├── test_vol_h5_new/
│ ├── test_h5_new/
ACDC
以上下载链接为预处理后的数据,将ACDC数据集下载后并放到data/ACDC下面
使用说明
2D 医学图像分割
训练
启动2D模型的训练:
cd 2d python train.py --args.model <网络名称>
测试
训练完成后,测试2D模型:
python test.py --args.model <网络名称> --pretrained_pth <权重路径>
3D 医学图像分割
训练
使用3D模型进行训练:
注意:此部分参考自3DUX-Net,修复了monai的裁剪问题,更多的细节请移步原作者主页 3DUX-Net。
cd 3d python main_train.py --root <root_folder> --output <output_folder> \ --dataset flare --network 3DUXNET --mode train --pretrain False \ --batch_size 1 --crop_sample 2 --lr 0.0001 --optim AdamW --max_iter 40000 \ --eval_step 500 --gpu 0 --cache_rate 0.2 --num_workers 2
测试
测试3D分割模型:
python test_seg.py --root <path_to_image_folder> --output <path_to_output> \ --dataset flare --network 3DUXNET --trained_weights <path_to_trained_weights> \ --mode test --sw_batch_size 4 --overlap 0.7 --gpu 0 --cache_rate 0.2
模型集成
2D 网络
2d/other_networks/ 文件夹中包含了各种2D分割模型,如U-Net、Swin UNet等。您可以根据需要修改或添加新的网络。
3D 网络
3d/networks/ 文件夹用于存储3D模型,如3D U-Net、nnFormer等。您可以在此处扩展新的3D模型架构。
可视化代码在相邻仓库
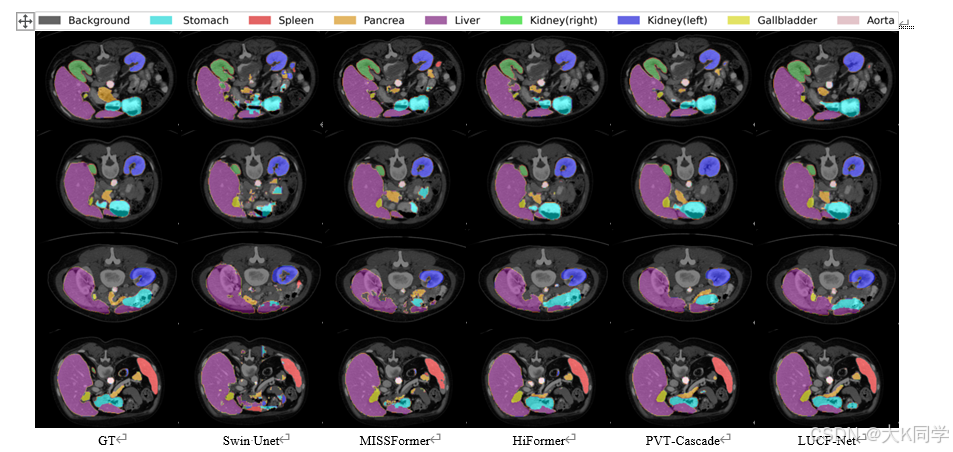
代码有问题欢迎小伙伴们联系我,后续会上传半监督的分割框架
GitHub - aixiaobaikkk/DeepMedSeg

火山引擎开发者社区是火山引擎打造的AI技术生态平台,聚焦Agent与大模型开发,提供豆包系列模型(图像/视频/视觉)、智能分析与会话工具,并配套评测集、动手实验室及行业案例库。社区通过技术沙龙、挑战赛等活动促进开发者成长,新用户可领50万Tokens权益,助力构建智能应用。
更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容





所有评论(0)