【GWAS】 2-全基因组分析可视化-网站部分
本文介绍了全基因组关联分析(GWAS)的基本概念和常用可视化网站。GWAS通过分析单核苷酸多态性(SNP)与表型的关联来定位致病变异,利用连锁不平衡原理进行标记。重点推荐了6个GWAS数据库网站:BnaGWAS(自带示例数据)、easyGWAS(含多物种数据)、GWAS Atlas(基于英国生物库)、AraGWAS Catalog(拟南芥专用)、GWAS Catalog(人类数据)和GWAS Ce
2-全基因组分析可视化-网站部分
GWAS数据结构
SNP概念
首先,全基因组关联分析(GWAS) 的核心分析对象是 单核苷酸多态性(SNP)。
SNP 是指在基因组水平上,由单个核苷酸的变异引起的 DNA 序列多态性。
简而言之: 它是指 DNA 序列中,某个特定位点上,一个碱基(A、T、C 或 G)被另一个碱基替换的现象。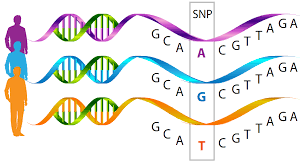
变异类型: SNP 主要由单个碱基的转换(purine → \to → purine 或 pyrimidine → \to → pyrimidine)或颠换(purine → \to → pyrimidine 或反之)导致,但也可能由单个碱基的插入或缺失引起。
频率与分布: 这些可变的位点(SNP)在人类全部遗传信息总量中占比很小,大约占 1% 左右。
GWAS概念
GWAS (Genome-wide association studies) 是一种强大的研究方法,专门用于系统性地识别与统计分析群体中 单核苷酸多态性(SNP)。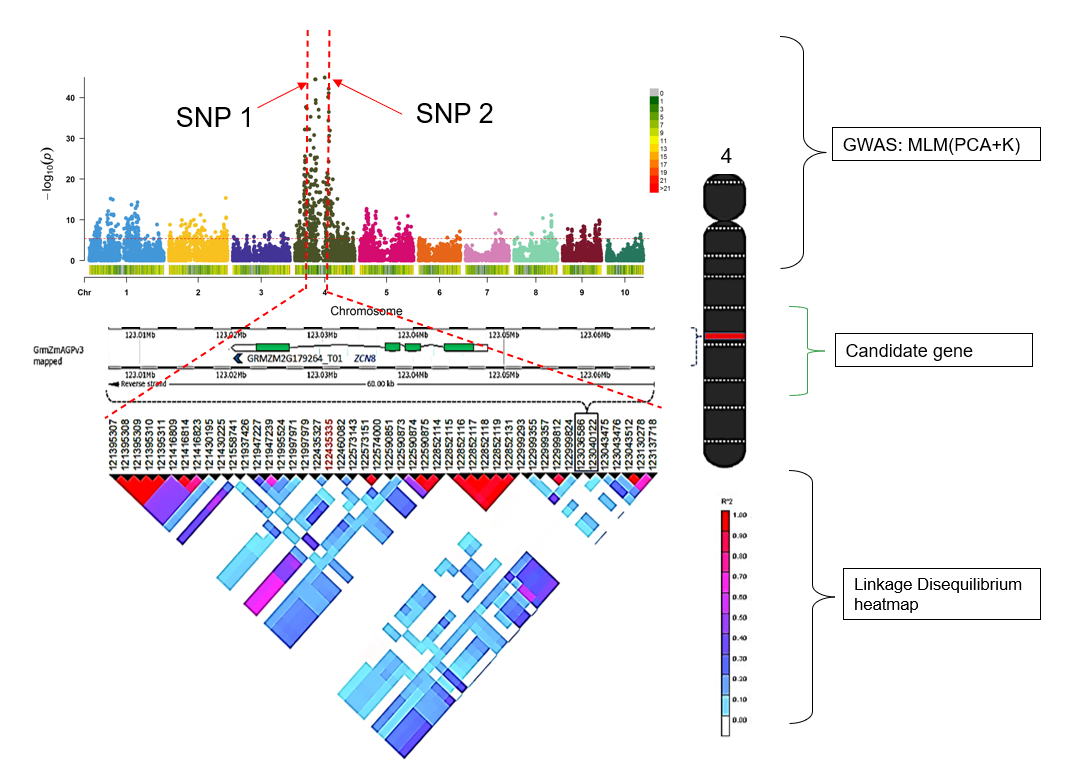
核心思想:通过对大量个体的全基因组SNP分型,并将其与特定的表型(如疾病、身高、药物反应等)进行比较分析,从而定位出与目标性状存在显著关联的基因组位点。
技术基础: GWAS 工作的关键在于 连锁不平衡(Linkage Disequilibrium, LD) 原理。
LD 原理: 这种原理指出,基因组上相邻近的遗传变异位点倾向于作为一个整体共同遗传。因此,GWAS 可以通过分析群体中常见的 “标记SNP”,来间接捕获并定位附近真正的 “致病变异” 。
GWAS绘图网站
BnaGWAS
使用网站自带示例数据进行作图。
曼哈顿图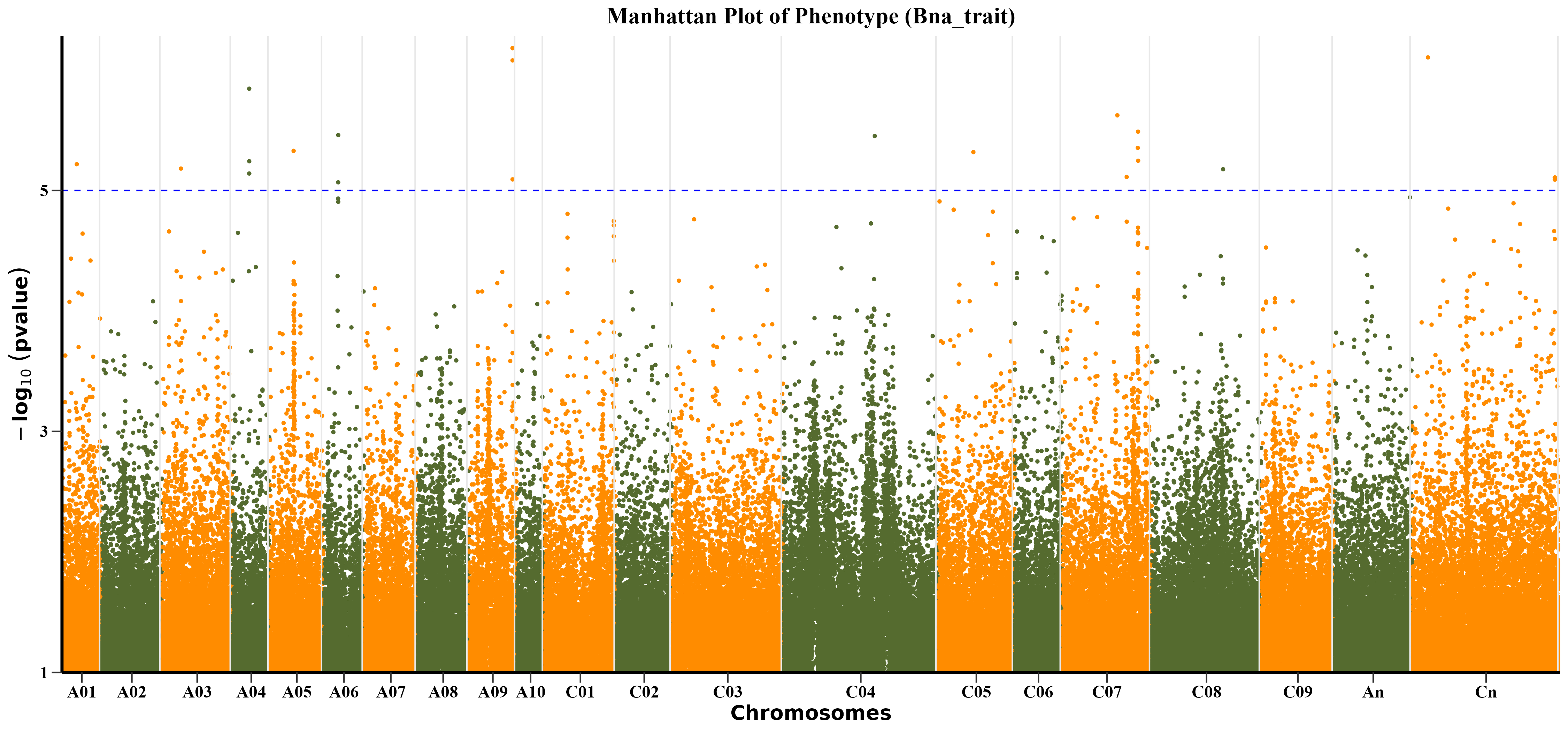
QQ图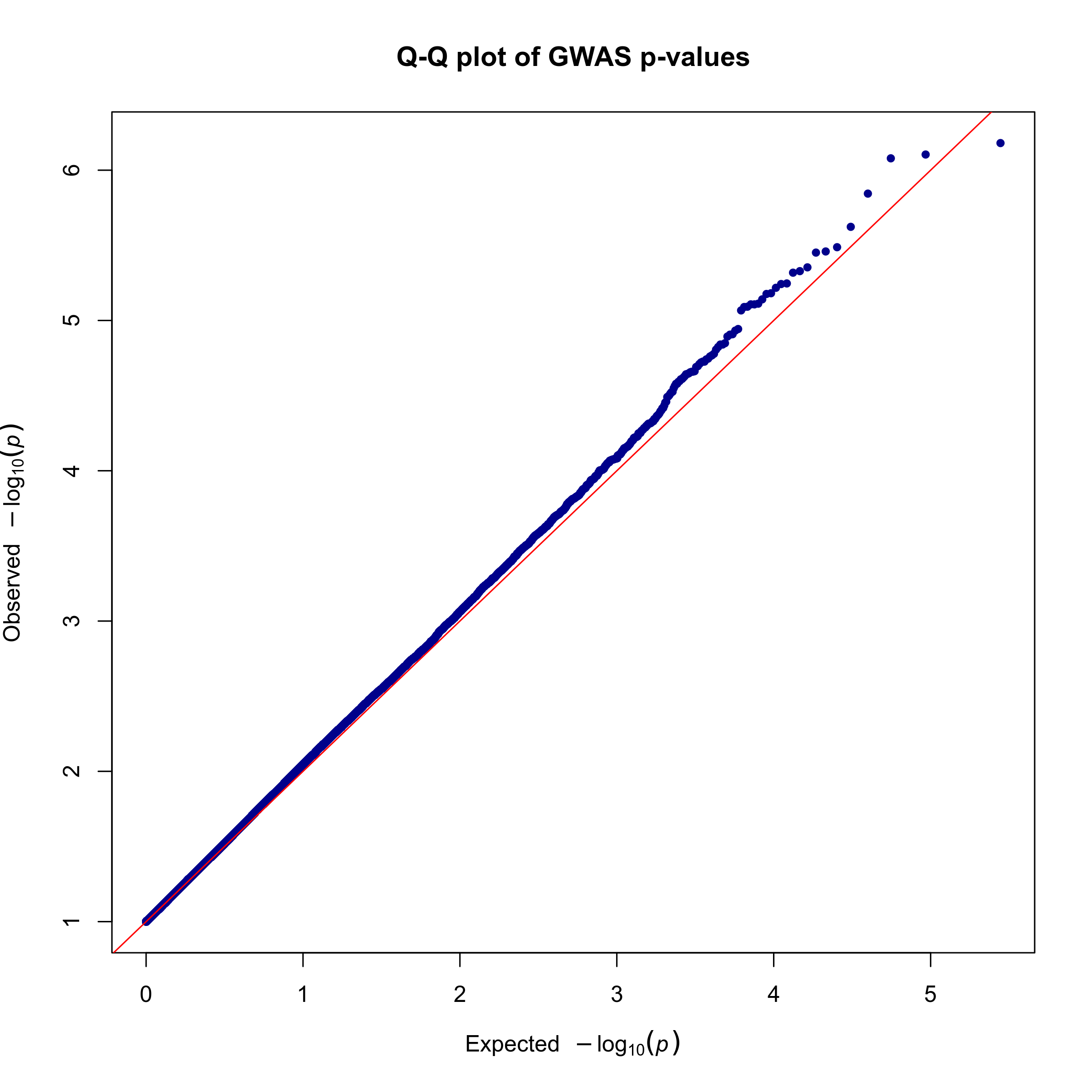
easyGWAS
easyGWAS是2016年发表再Plant cell上的Web网站(https://doi.org/10.1105/tpc.16.00551)。
包含拟南芥、果蝇线虫等模式植物以及多个物种的公开数据。
网站自带教程,使用时可以参考:https://easygwas.biochem.mpg.de/faq/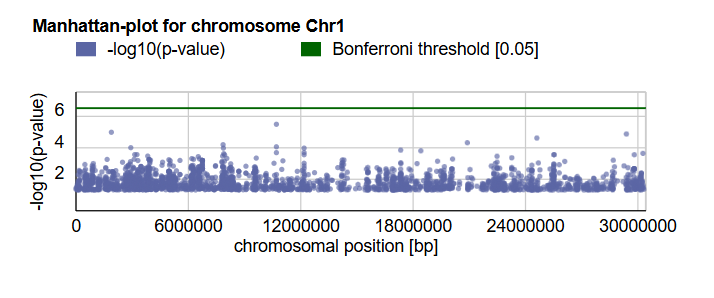
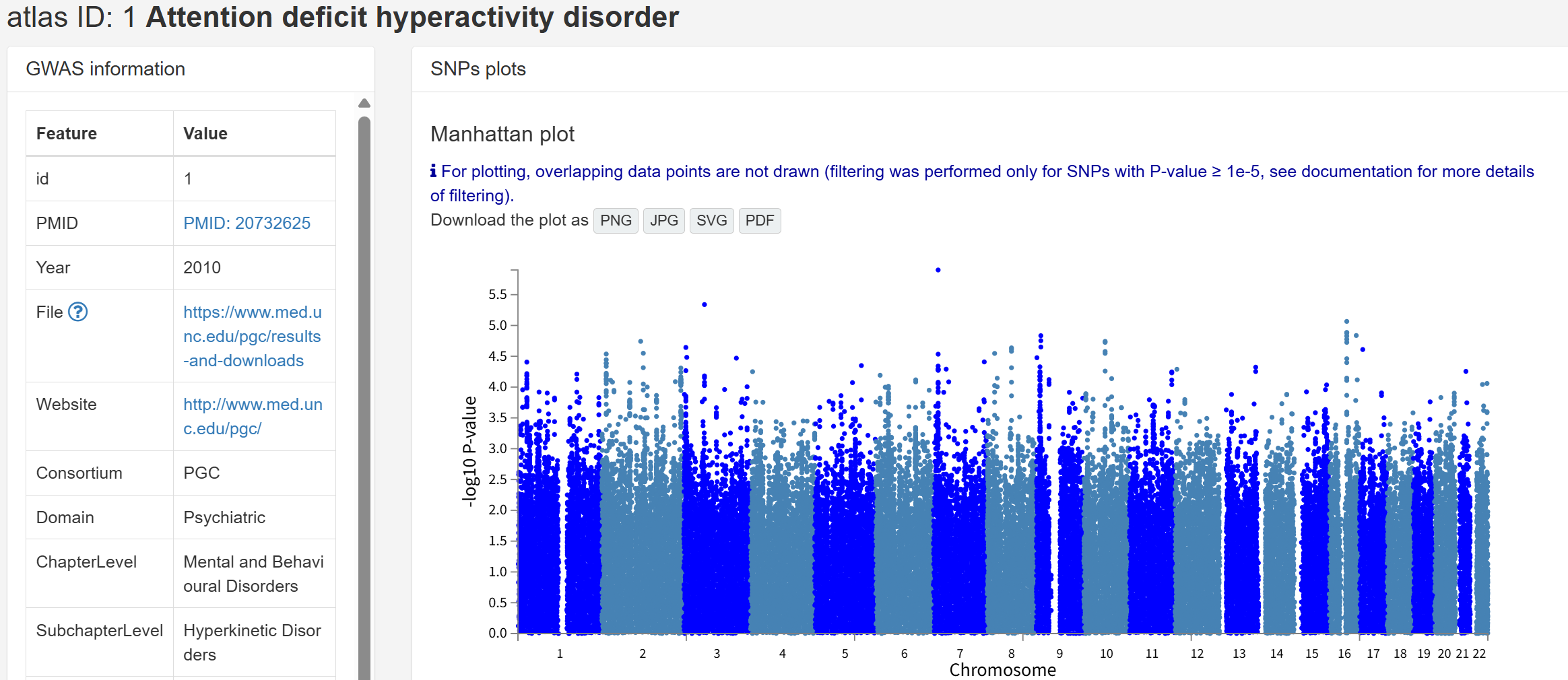
GWAS Atlas
GWAS Atlas是基于英国生物库数据建立的公开GWAS数据库,提供600项研究的曼哈顿图、遗传相关性等可视化结果,支持数据下载,并鼓励用户补充未收录的公开数据。
AraGWAS Catalog
AraGWAS Catalog 是一个公开、手动管理、收录拟南芥标准化全基因组关联分析(GWAS)结果的数据库,支持对研究、表型和基因进行检索筛选,并提供统一方法重新计算所得的元信息。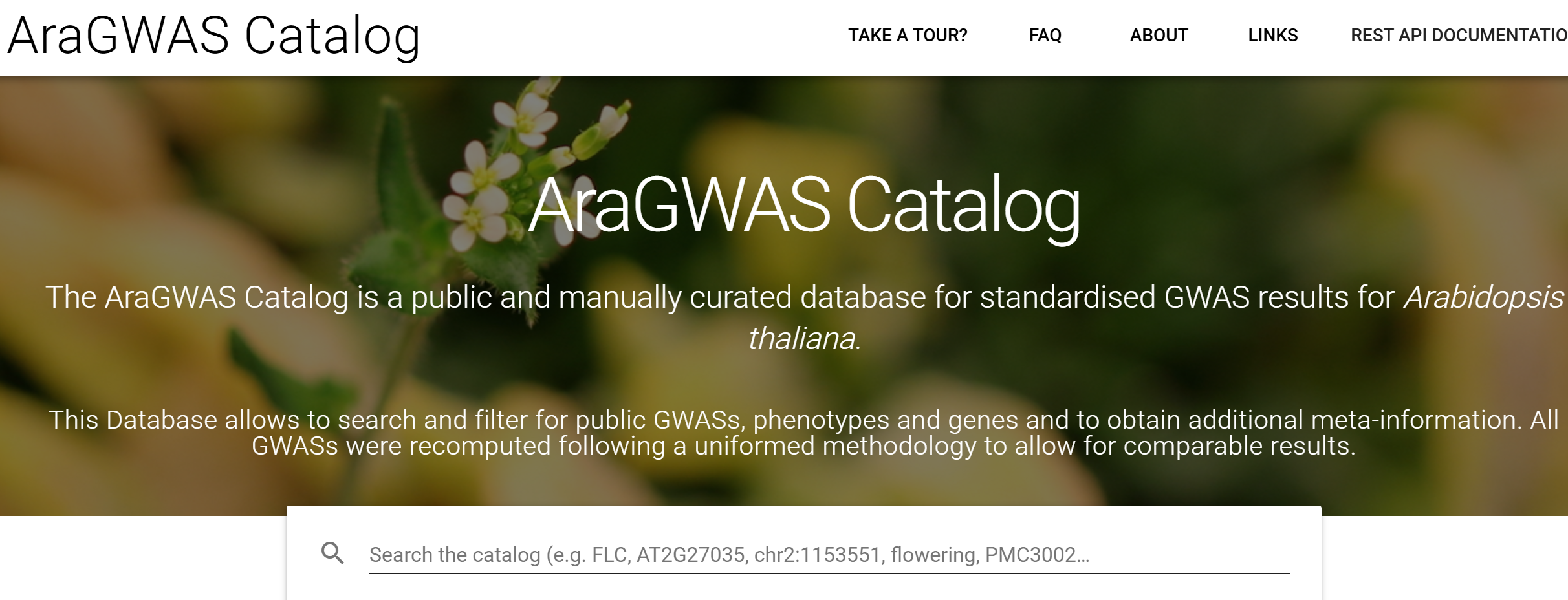
GWAS Catalog
GWAS Catalog是一个收录近6000项研究、5000多种表型、约40万个显著位点的人类GWAS数据库。支持数据查询、交互浏览与上传,2022年更新于《Nucleic Acids Research》。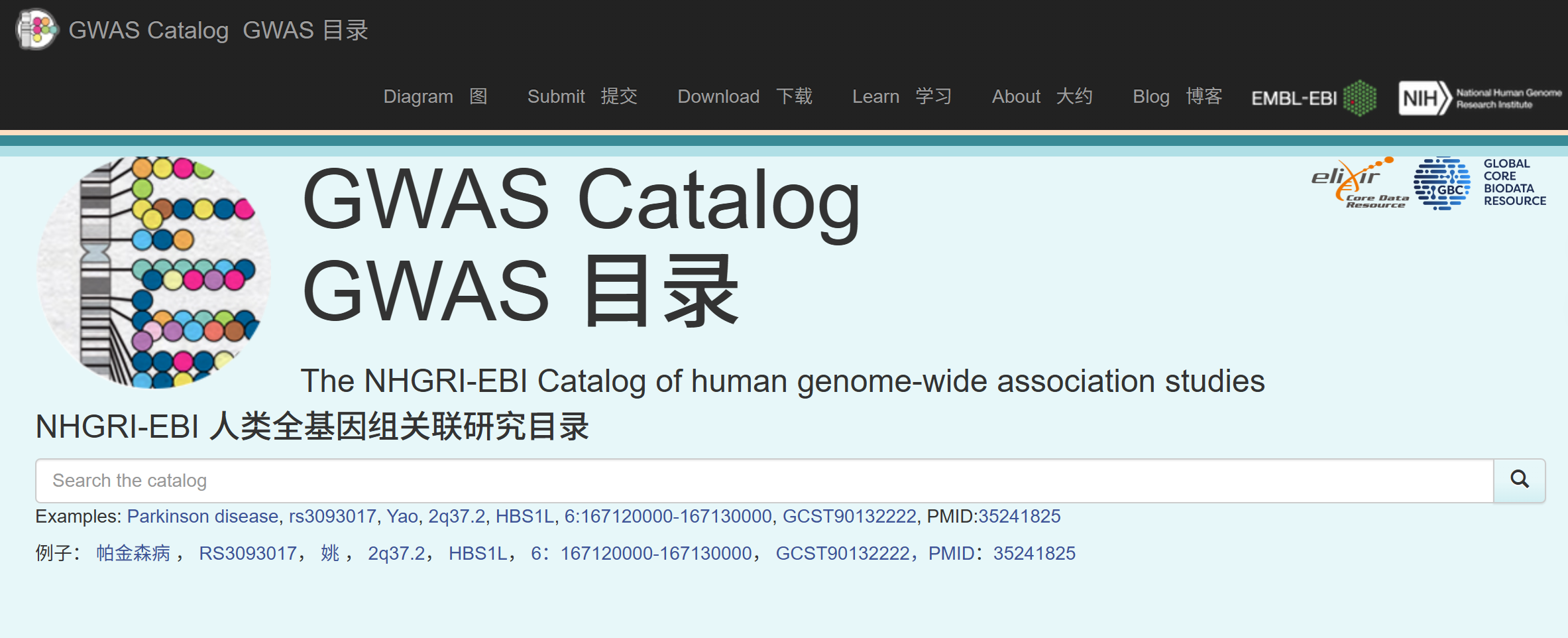
GWAS Central
GWAS Central是一个汇总遗传关联研究结果(如等位基因频率和显著性数据)的数据库,数据源于公共项目及社区提交。它以公共变异数据库为基础,按研究实验、样本组和表型进行组织,仅包含汇总数据,不涉及个体基因型或表型信息。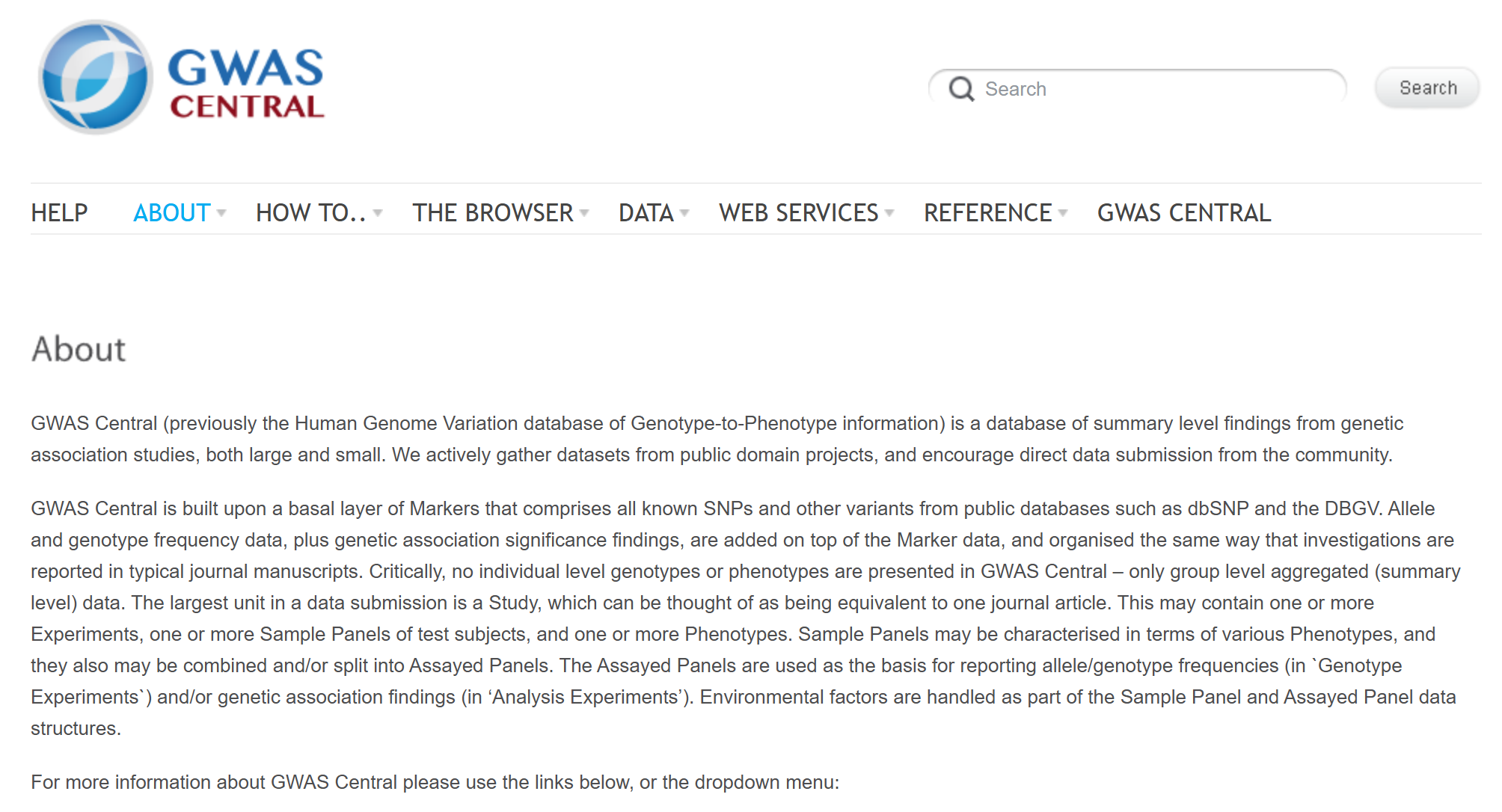
推荐博客
在学习GWAS相关课程和资料时,找到一个研究GWAS的大佬写的博客:gwaslab
后续复现
后续我们将复现 Liu, Y., Wang, L., Mao, S. 等人在《Scientific Reports》上发表的研究“Aegilops tauschii 中29个形态性状的全基因组关联分析”(2015, DOI: https://doi.org/10.1038/srep15562)。

火山引擎开发者社区是火山引擎打造的AI技术生态平台,聚焦Agent与大模型开发,提供豆包系列模型(图像/视频/视觉)、智能分析与会话工具,并配套评测集、动手实验室及行业案例库。社区通过技术沙龙、挑战赛等活动促进开发者成长,新用户可领50万Tokens权益,助力构建智能应用。
更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容






所有评论(0)